3d基因组技术(3D类脑组织中进行基因编辑和谱系追踪的新方法)
本研究报道的这项高通量筛选技术已经被用来在3D细胞培养中筛选与神经发育性疾病有关的基因,该技术揭示了一种罕见的被称为小头畸形症可能涉及的发病机制。与自闭症、精神分裂症和癫痫症等神经发育性障碍有关的这些基因,都可以通过该技术筛选出来。
基因成千上万种的变异方式增加了个体受神经系统严重紊乱影响的风险,例如:出现大脑发育不良1-3。了解这些基因变异所产生的影响,可以揭示疾病的发病机制,同时还有助于发现可能存在的新的药物靶点。但是单独对每种基因变异进行研究是一个缓慢且艰难的过程。

除此之外,人类大脑的复杂性也使得此类研究的进行变得愈发困难。Esk等人4在《科学》杂志上发表了一篇论文,文章中概述了一种被称为高通量体外筛选的方法,此种方法有望克服上述研究阻碍。随后,作者利用他们所描述的这种筛选技术,对一种被称作小头畸形症的罕见发育状况的相关基因进行了分析。
患有小头畸形症患者的大脑——特别是大脑的外部,即大脑皮层——比普通大脑要小得多1-3。之所以出现这样的现象原因有二,由于神经干细胞增殖的减少或是由于大脑发育过程中细胞死亡的增加。小头畸形症常伴随着一系列的基因和遗传综合征1-3。
若要确定这些基因是否确实与小头畸形症有关,需要用到高通量技术,但是现有的筛选方法有其局限性。举例来讲,那些使用动物来验证效果的高通量技术研究可能是存在问题的,因为在不同的动物模型中,等效基因突变可能会产生不同的影响5,并且与神经发育障碍相关的基因在不同物种中的表达可能是不同的。2D细胞培养系统不能完全捕捉3D组织中不同类型神经元和神经胶质细胞之间的交互作用等过程,而这些过程对于正常细胞的增殖来讲至关重要。
但是被称为类器官的3D细胞培养系统,可以通过干细胞生成类似大脑的结构,这也为克服使用动物模型和2D细胞培养所面临的问题提供了一种强有力解决方案6。类器官会形成类似大脑的组织,可以重现人类从妊娠早期到中期皮质发育过程中关键细胞和基因的表达特征6,7。
然而,以3D形式模拟复杂组织对于高通量筛选来讲是一把双刃剑。不同细胞群生长速率的可变性,使得我们难以确定细胞存活或增殖所存在的差异是由于实验的性质还是由于类器官固有的变异性。
Esk等人试图通过将脑类器官与一种基于基因编辑的新技术CRISPR-Cas9相结合的方法,来克服上述问题。在CRISPR-Cas9技术中,引导性RNA(gRNA)被设计成能够与即将被编辑的DNA序列结合的形式。Cas9酶随后与gRNA结合并使DNA链断裂。通常情况下,这种DNA链断裂会被细胞错误地修复,而修复的结果可能导致基因被“敲除”,也就是说我们感兴趣的基因将不再被翻译成功能性蛋白质。
Esk及其同事针对173个与小头畸形症有关的基因设计了相对应的gRNA,并且使用一种称为谱系条形码的独特DNA序列对每个gRNA进行了标记。随后,他们用这些条形码gRNAs感染了干细胞。每个接收到给定gRNA的细胞及其所有后代携带的条形码都是相同的,这方便了我们对细胞谱系进行识别。将干细胞在3D培养基中培养40天形成一个脑类器官,随后提取并汇集所有类器官细胞中的DNA。
筛选的目的是计算每个感兴趣基因的谱系条形码的数量,并将该数值与未发生任何基因突变的对照gRNA的谱系条形码数量进行比较。如果与对照gRNA谱系条形码的数量相比,候选基因的谱系条形码发生了损耗,则表明该基因是细胞正常增殖所必需的。但是,在分析过程中我们很难检测到数量较少的谱系条形码(即来自小谱系的条形码)。这就需要我们设定更高的分辨率,从而确保能够检测到中度细胞丢失的情况(而中度细胞丢失这是小头畸形症的典型特征)。除此之外,正如上文所述,在一个3D脑类器官中,基线谱系大小的变化是非常大的。
为了解决这些问题,Esk等人在DNA处理过程中为每个细胞添加了第二类条形码,从而对谱系中每个细胞的DNA分别进行了标记(图1)。对细胞进行单个的条形码标记,使得研究人员能够精确地追踪每个谱系中究竟有多少细胞。
双重条形码标记共同解释了类器官细胞生长的变异性(每个基因可以使用多个gRNAs,从而可以更可靠地检测到细胞变异的趋势),同时对每个细胞进行条形码标记使得我们检测小谱系的细胞增殖变化成为可能,这也更方便我们对基因缺失导致的中度细胞丢失的情况进行识别。该研究团队将他们所使用的这种技术命名为,异质组织中以细胞分辨率进行的CRISPR谱系追踪(CRISPR-LICHT)。
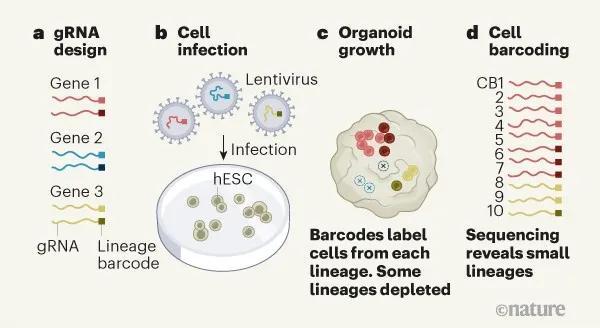
图1 | 基因编辑和谱系追踪方法——CRISPR-LICHT。Esk等人利用CRISPR-LICHT方法对基因突变是如何改变大脑细胞数量这一问题进行了分析。a、研究者设计了引导性RNA(gRNA)分子,每个分子与173个感兴趣的基因中的一个进行结合。并且他们使用一种被称为谱系条形码(LB)的独特DNA序列对每个gRNA分子进行了标记。b、然后将每个gRNA包装成慢病毒(为了看起来更简洁,这里只显示了三个gRNA包装成的慢病毒),并使用慢病毒池感染人类胚胎干细胞(hESCs)。c、随后将细胞置于3D培养基中,并催生Cas9酶(该酶的目标是gRNAs,它能够使gRNAs删除每个受感染细胞中的基因;图中未显示)。细胞将被培养至40天,从而生成复杂的3D脑类器官。有些谱系细胞生长迅速,而另外一些谱系则大量减少(杂交)。d、随后对类器官进行收集并选择其中含有Cas9的细胞(图中未显示)。对这些细胞中的DNA进行扩增,并添加第二种独特的DNA条形码(细胞条形码,CB)。然后按照gRNA和条形码对这些DNA进行测序。LBs(谱系条形码)是我们能够精确测量每个谱系的大小,CBs(细胞条形码)增加了我们挑选小谱系的能力。如果含有某一基因gRNAs的谱系一直很小(这里可以参看基因2和基因3),那么该基因很可能在脑细胞增殖中发挥作用。(图源:nature)
研究人员对173个被认为引起患小头畸形症风险的基因进行了谱系条形码缺失排序,并对排名前32的基因进行了更为详细的检验。他们利用混合策略研究了这32个基因在调节细胞增殖中所发挥的作用,该混合策略指的是,他们从控制细胞和经过基因编辑缺失了某一基因的细胞结合体中培养出类器官,并分析了完全长成的类器官中控制细胞与编辑细胞的比率。如果发现编辑过的细胞数量显著减少,则表明基因缺失导致了细胞增殖的减少。在被研究的32个基因中,有25个被发现与细胞增殖有关。
最终,Esk等人集中研究了该基因集合中的一个特定基因,该基因主要负责的是编码即时早期反应3相互作用蛋白1(IER3IP1)。当细胞被敲除该基因时,产生的类器官比对照类器官小。对缺乏这种基因的类器官的分析结果表明,该基因似乎是用来调节未折叠蛋白反应的(即细胞内一种称为内质网的细胞器对压力的反应,这种反应会导致蛋白质合成的减少)。
使用一种能够恢复蛋白质合成的被称为整合应激反应抑制剂的小分子对细胞进行处理,会使类器官中神经前体细胞的大小和组织恢复正常。这些发现是非常有趣的,因为它们将压力诱导的未折叠蛋白反应(该反应此前被认为与Zika病毒相关的小头畸形症8有关)与患有疾病的遗传因素联系在一起。这些实验强调了CRISPR-LICHT技术在研究神经发育障碍的机制方面的巨大潜力。
尽管Esk及其同事在研究中主要关注的是与小头畸形症相关的基因,但是他们所使用的高性价比且省时的方法将广泛适用于其他神经发育障碍。发育性脑部疾病基因数据库(go.nature.com/3mezn8d)列出了约600个基因,这些基因的缺失与自闭症、精神分裂症和癫痫症等神经发育性障碍有关。现在,这些基因都可以通过CRISPR-LICHT技术筛选出来。
这种方法也可以通过调整基因来改变而不是消除基因表达,从而对神经发育障碍中低表达或过度表达的基因进行研究。除此之外,CRISPR-LICHT技术中所使用的双条形码技术也可以被用于创建和跟踪在遗传上有所区别的细胞群体,从而模拟大脑发育过程中细胞间遗传变异所产生的影响9。最后,这种方法可用于分析与癌症相关的基因。
但是,这一方法也存在一定的局限性。CRISPR-LICHT技术最适用于与早期发育或细胞增殖相关的研究。本研究中的大脑类器官的生长时间为40天,这模拟的是人类在妊娠早期的大脑发育,因此没有考虑后期发育成熟的神经元和胶质细胞。使用更长的分化时间,将会使周转期增加。
除此之外,当前的类器官生成方案可能激活细胞的应激路径,从而损害细胞类型规格及其准确性10。最后,并不是所有的皮层都是一样的,不同的皮层区域有不同的细胞类型和连接,起着不同的作用。我们需要设计出更为精确的方案来对这种复杂性和区域化进行模拟。
除临床研究外,CRISPR-LICHT平台还可用于研究健康人脑进化过程中的遗传变化及机制。有证据表明,人类皮层的扩张和折叠取决于人类早期神经元发育的特性11-13。使用大脑类器官来筛选人类特有的或者在人类中表现出独特活动模式的基因和其他DNA元素14,这可能是在人类神经发育的背景下,将基因与人类特征联系起来的一种强有力的方法。
参考信息:
1.Woods, C. G. Curr. Opin. Neurobiol. 14, 112–117 (2004).
2.Dixon-Salazar, T. J. & Gleeson, J. G. Ann. NY Acad. Sci. 1214, 156–167 (2010).
3.Jayaraman, D., Bae, B.-I. & Walsh, C. A. Annu. Rev. Genom. Hum. Genet. 19, 177–200 (2018).
4.Esk C. et al. Science 370, 935–941 (2020).
5.Liao, B.-Y. & Zhang, J. Proc. Natl Acad. Sci. USA 105, 6987–6992 (2008).
6.Eiraku, M. et al. Cell Stem Cell 3, 519–532 (2008).
7.Mariani, J. et al. Proc. Natl Acad. Sci. USA 109, 12770–12775 (2012).
8.Gladwyn-Ng, I. et al. Nature Neurosci. 21, 63–71 (2018).
9.McConnell, M. J. et al. Science 356, eaal1641 (2017).
10.Bhaduri, A. et al. Nature 578, 142–148 (2020).
11.Heide, M. et al. Science 369, 546–550 (2020).
12.Fiddes, I. T. et al. Cell 173, 1356–1369 (2018).
13.Suzuki, I. K. et al. Cell 173, 1370–1384 (2018).
14.Sousa, A. M. M., Meyer, K. A., Santpere, G., Gulden, F. O. & Sestan, N. Cell 170, 226–247 (2017).
原文作者:Adriana Cherskov & Nenad Sestan
编译作者:UTCS(brainnews创作团队)
校审:Simon(brainnews编辑部)
,免责声明:本文仅代表文章作者的个人观点,与本站无关。其原创性、真实性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容文字的真实性、完整性和原创性本站不作任何保证或承诺,请读者仅作参考,并自行核实相关内容。文章投诉邮箱:anhduc.ph@yahoo.com






